- Barajar
ActivarDesactivar
- Alphabetizar
ActivarDesactivar
- Frente Primero
ActivarDesactivar
- Ambos lados
ActivarDesactivar
- Leer
ActivarDesactivar
Leyendo...
Cómo estudiar sus tarjetas
Teclas de Derecha/Izquierda: Navegar entre tarjetas.tecla derechatecla izquierda
Teclas Arriba/Abajo: Colvea la carta entre frente y dorso.tecla abajotecla arriba
Tecla H: Muestra pista (3er lado).tecla h
Tecla N: Lea el texto en voz.tecla n
![]()
Boton play
![]()
Boton play
![]()
21 Cartas en este set
- Frente
- Atrás
|
tenim 5 estrategies de silenciament de la funció gènica, quines??
|
1. oligonucleotods antisentit
2. RNA interferència 3. short hairpin RNAs o shRNAs 4. edició del genoma: zinc finger nucleases, tales transcription activator-like efectors o CRISPR/Cas9 5. estrategua Exon skipping |
|
silenciamemt amb oligonucleotids antisentit.
quantes bases el formen? direcció seq? pq silencia?? |
són polimers sintetics formats x desoxint o ribont modificats amb una longitud de 15-20bases.
seq 3'-5', complementaria a una dr les cadenes de mRNA. es silencia pq forma una estructura de doble cadena i o no es tradueix o les endonucleases el degraden i x tant hi haurà menys producció de proteïna. |
|
quin és el mecabksme da ció deks oligo antis x silenciar? poden jnir-se a exons i introns
|
EXONS: Doble cadena oligontas impedeix traducció i fa q endonucleases degradin el mRNA
INTRONS: no es farà l'splicing. |
|
2. RNA interferencia. a quin nivell impedeix l'expressió gènica
|
- traducció
- transcripció (la dificulta( |
|
RNAint. com funciona??
|
quan un virus/bact infecta una cell com a defensa es sintetitza dsRNA (RNA de doble cadena) i amb la proteina DICER es trenquen a siRNA (RNA +petits i de cadena simple).
si els siRNAs són complementaris a una det. seq de mRNAs, es forma un complex DICER, siRNA i proteina RISC q impedeix la traducció del gen |
|
disseny d'un siRNA
1. longitud 2. 4 regions a evitar 3. repeticions si o no? 4. analisis q cal fer. 5. disseny controls +o-? |
1. longitud: 21nt.
2. 4 regions a evitar: compreses entre el 50-100bp del codo inici i de l'stop, reg introniques, amb molt o poc GC (30-60% b), repeticions de 4 o + bases. 3. repeticions si o no? 4. analisis q cal fer. blast. 5. disseny controls +o-? - |
|
com podem comprovar l'efectivitat dels siRNAs?
|
1. quantificar l'RNAm: fent una RT-PCR (a punt final no valdria)
2. quantificar la proteïna:fent un western-blot amb un anticos 1a dirigit a la proteina o un anticos 2a dirigit a l'Ac 1a i marcat |
|
3. tec shRNAs o short hairpin RNAs. quin és el seu mecanisme? quin avantatge tenen respecte els siRNAs?
|
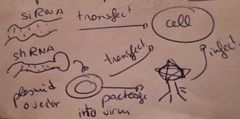
mateix mecabisme que els siRNAs però s'introdueixen a les cells en un vector i aixi la seva expressió pot ser transitoria, induïble o permanent.
elnplasmidi té una seq de DNA q transcriu shrna (cadena simole xo q té 2 seq complementaries sobre elles mateixes i formaran un loop, en el q hi ha dianes de restricció q acabaran formant els siRNAs. |
|
pegues i inconvenients dels siRNAs
|
activació de la resposta immune i inhibició transitoria
|
|
les 3 estrategies d'edició del genoma x una resposta permanent són..? en què es basen??
|
1. zinc finger nucleases
2. tales transcription activator-like efectors 3. CRISPR/Cas9 es basen l'ús d'endonucleases q tallen la doble cadena de DNA, els dits de ZN i Tales són proteines q detecten de manera molt especofica una seq de nt i serveixen x crear knock-out i knock-in |
|
Zn Finger:
quants factors de transcripció hi ha?? a quin domini catalitic s'uneix?? |

3 factors q s'uneixen al domini catalitic de ma proteina Fok I q és una endonucleasa
|
|
Zn Finger:
quina estructura tenen?? què detecten?? qui fa el tall a quants nt?? com funciona FokI |
estructura ZnF: 31aa rics en cisteines i histidines que detecten de manera molt fidel 3 nt.
el tall el fan 3 dits de Zn com a minkm que determinaran una seq de 9nt és a dir els dits de Zn reconeixen una seq de 9nt i FokI fa el tall. fokI funciona com a dimer, calen 2 x fer el tall, cadascuna s'uneix a una cadena |
|
TALES:
Origen?? format x?? qui fa el tall?? |
prove del patogen de les pantes Xanthomonas sp. són proteines d'unió al DNA especifiques de lloc amb un domini d'unió molt conservat format de 33-35aa
el peptid TALE es fusiona al domini catalitic FokI que tallarà la cadena de DNA, igual q en ZNFinger cal un dimer per fer el tall. |
|
Cert o fals: els dominis catalutics de la prpteïna FokI aniran units als mateixos pèptids FokI
|
fals, pq FokI té els pèptids Tale a esquerra i dreta units a diferebts punts de la mateixa cadeina
|
|
mecanisme d'acvió de tale i znf x knock-out i -in
|
-out: la cell detecta q hem tallat i intenta reparar introduint nt a l'atzar, lo + probable és q es perdi el marc de lectura-> silenci.
-in: afegeix un fragment de DNA de doble cadena q tingui els 2 extrems seq de RecomHomo. |
|
cicles q pateix la cell quan és infevtada x un virus
|
1- CICLE LITIC: es recircula el DNA del virus fent plasmidis i creant copies del material generic del.virus i les seves proteines trencant la cell hoste
2-CICLE LISOGÈNIC: DNA s'incorpora al cromosoma de la cell hoste i llavors forma els CRISPR arrays. es poden sintetitzar proteines i crear virions xo el material es queda permanentment. la cadena de RNA es fragmentarà en trossos petits i s'unirà al material genetic dl virus si hi ha una nova infecció i s'activaran les proteines Cas9 x evitar la nova infecció. |
|
que necessitem pl sistema CRISPSR
|
1- gen que volem editar
2- cadena de RNA guia complementaria a la seq del gen on volem fer el tall. A la q afegirem el fragment SCAFFOLD q s'unirà especificament a la proteina Cas9 (endonucleasa) |
|
que necessita la proteina Cas9??
|
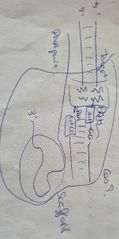
la proteina Cas9 necessita la seq PAM (seq diana per fer el tall)
-seq PAM cadena codificant: 1nt+2G -seq PAM cadena comple. 1nt+2C |
|
mecanisme CRISPSR
|
el domini Scaffold q està unit a la cadena guia atraurà a la proteina Cas9 q detectarà la diana PAM i tallarà la doble cadena.
|
|
a antatge CRISPR en front Tales i ZnF
|
crispr actua x moltes especies, els altrea dos x poques.
|
|
estrategia exon skipping
malaltia?? exo afectat? estrategia? problema de lestrategia? |
la malaltia distrofia muscular de Duchenne està lligada al cromosoma X, el gen de la proteina distrofina té una mutació a l'exó51 q provoca la terminació prematura de la proteï a, no afecta ni a l'estructura ni a la funció.
x evitar l'ecpressió de l'exó51 usem oligont antisentit però no és permanent. |