- Barajar
ActivarDesactivar
- Alphabetizar
ActivarDesactivar
- Frente Primero
ActivarDesactivar
- Ambos lados
ActivarDesactivar
- Leer
ActivarDesactivar
Leyendo...
Cómo estudiar sus tarjetas
Teclas de Derecha/Izquierda: Navegar entre tarjetas.tecla derechatecla izquierda
Teclas Arriba/Abajo: Colvea la carta entre frente y dorso.tecla abajotecla arriba
Tecla H: Muestra pista (3er lado).tecla h
Tecla N: Lea el texto en voz.tecla n
![]()
Boton play
![]()
Boton play
![]()
55 Cartas en este set
- Frente
- Atrás
¿ Qué es una diana biológica?
|
Es una macromolécula endógena al organismo que interacción con una molécula pequeña para generar una respuesta biológica.
|

Significado de sitio de unión
|
Es la fracción estructural de una macromolécula cuya interacción con una molécula endógena o externa se traduce en una respuesta biológica.
|
¿ Qué es un ligando?
|
Un compuesto químico que se une a un sitio diana el cual puede ser un neurotransmisor, hormona, fármaco, una molécula bioactiva, entre otras.
|
Significado de Agonista
|
Sustancia endógena, fármaco o molécula bioactiva que interactua con un receptor e inicia una respuesta farmacológica.
|

Significado de Antagonista
|
Compuesto químico que se opone al efecto fisiológico de otro, bloqueando al receptor y evitando que haya una actividad biológica.
|
Tipos de dianas biológicas
|
Macromoléculas:
Protéicas ° Proteínas No protéicas ° Lípidos ° Ácidos nucléicos ° Carbohidratos |
¿ Qué es un ligando?
|
Cualquier molécula que se une al blanco molecular.
|

Ajuste Inducido
|
Es cuando el ligando cambia su conformación para poder unirse al sitio de unión.
"Cambios en el ligando que provocan, promueven o inducen el cambio". |
Tipos de dianas biológicas
|
° Canales iónicos
° Acoplados a proteínas G ° Ligados a cinasas ° Nucleares |
Interacciones reversibles
|
Se presenta en la mayoría de los fármacos utilizados en el área clínica
|

Interacciones irreversibles
|
Se presenta en los agentes alquilantes, beta-lactámicos y pirazoles. Caracterizada por al menos un enlace covalente entre el ligando y el receptor.
|

Interacciones iónicas
|
° Consideradas las de mayor fuerza
° Interacciones iniciales más importantes ° Ocurren más en ambientes hidrofóbicos |

Puentes de hidrógeno
|
° Interacciones más débiles que las electrostáticas y más fuertes que las de Van der Waals
° Donador de enlaces de hidrógeno ° Aceptor de enlaces de hidrógeno |

Interacciones de Van der Waals
|
° Interacciones débiles
° Ocurren entre las regiones hidrofóbicas del fármaco y la diana ° El fármaco se debe encontrar cerca del sitio de unión para que la interacción ocurra |
Interacciones dipolo-dipolo
|
° Ocurre cuando el fármaco y el sitio de unión tiene momentos dipolo
° Los dipolos se alinean entre sí y ocurre la unión |
Interacciones ión-dipolo
|
° Ocurren cuando la carga de una molécula interacciona con el momento dipolar de otra
° Son un poco más fuertes que las interacciones dipolo-dipolo |
Interacciones dipolo inducido
|
° Ocurren cuando la carga de una molécula induce el dipolo de otra molécula
° Generalmente ocurre entre el ion amonio y un anillo aromático |
Significado de energía de solvatación
|
Existen moléculas de agua que rodean al ligando, cuando estas moléculas de agua se quitan tanto del ligando como del sitio de unión ocurre "Energy penalty", mientras que cuando interaccionan entre ellos ocurre "Enery gain".
|

Interacciones hidrófobas
|
° Ocurre cuando las regiones hidrofóbicas del fármaco y las de la diana no se encuentran solvatadas
° Como consecuencia hay un aumento en la entropía ° Beneficiosa en la energía de unión |

Enlaces covalentes
|
° Generalmente son interacciones irreversibles
Algunos ejemplos: son las penicilinas o algunos agentes alquilantes |
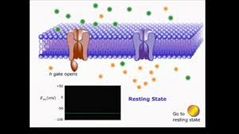
¿ Qué es un receptor?
|
Son proteínas unidas a la membrana que contienen un sitio externo el que se unen "mensajeros químicos" como neurotransmisores u hormonas, entre otras, que inducen un cambio conformacional en el mismo.
|
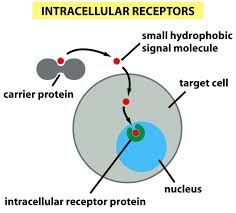
Receptores intracelulares
|
° No son transmembranales
° Se encuentran dentro de la célula ° Los ligandos deben de ser hidrofóbicos para que puedan atravesar la membrana celular |
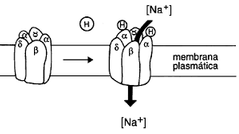
Receptores iónicos
|
Es un complejo de proteínas que atraviesan la membrana celular y permiten el paso de iones específicos como sodio, potasio, calcio y cloro.
Son complejos de subunidades de proteína |
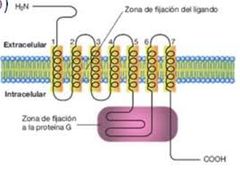
Receptores acoplados a proteínas G
|
El receptor transmite su mensaje vía una proteína de señalización llamada proteína G.
Es una proteína de 7 asas transmembranales Tiene dos sitios de unión: una parte extracelular y el otro sitio específico para la proteína G |
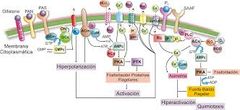
Receptores ligados a Cinasas
|
Actúan como receptores y como enzimas.
La unión del mensajero (ligando) en la parte extracelular activa a la enzima en la parte intracelular debido a que se llevan a cabo fosforilaciones. |
Inhibidores competitivos
|
Compiten con el sustrato natural por el sitio activo
|

Inhibidores NO competitivos
|
NO compiten con el sustrato natural por el sitio activo.
Se unen en sitios diferentes. |
Inhibidores reversibles
|
Interaccionan con el receptor con interacciones intermoleculares no covalentes.
|

Inhibidores irreversibles
|
Generalmente contiene grupos electrofílicos reactivos que forman enlaces covalentes.
|
¿Qué es una isoenzima o isoforma?
|
Se le denomina así cuando una misma enzima puede existir en diferentes conformaciones, en donde la reacción catalítica es la misma pero la composición de los aminoácidos es diferente.
|
Docking Molecular
|
Búsqueda del modo de unión preferido del ligando en un complejo ligando-receptor.
|
Funciones del Docking molecular
|
° Elucidar modo de unión de moléculas con actividad biológica
° Se pueden genera diversas hipótesis de la posible relación entre el modo de unión y la actividad de las nuevas moléculas. |
¿ Qué componentes se necesitan para realizar un docking?
|
1. La estructura del blanco molecular
2. La estructura del compuesto Obtenidas por diferentes métodos como cristalografía, resonancia magnética nuclear o modelado molecular, entre otras. |
¿Cuales son algunos pasos que se necesitan realizar para preparar los datos?
|
° Normalización de quimiotipos
° Realizar tratamientos en caso de tener sales ° Eliminar las estructuras duplicadas ° Verificar si existen formas tautoméricas ° Verificar los estados de protonación |

¿ Qué criterios se deben de considerar para realizar la elección de los programas de docking?
|
Todo depende de la investigación que estemos realizando, aunque algunos factores a considerar son:
° Precisión ° Velocidad ° Acceso a parámetros ° Costo-beneficio ° Duración de las licencias Nota*: Se recomienda siempre considerar más de un programa para que los resultados obtenidos sean más confiables. |
¿Como se pueden validar los protocolos del docking?
|
1. Se pueden reproducir las coordenadas del ligando en su forma cristalizada
2. Corroborar que el modo de unión concuerde con el mecanismo reportado 3. En el cribado virtual: corroborar que el docking score distinga las moléculas activas de las inactivas |
¿Cuáles son algunos pasos para el post-procesamiento de datos?
|
° Selección automatizada de hits
- Por orden relativo - Por score ° Eliminar los falsos positivos - Realizando un consensus docking - Realizando un consensus scoring - Por combinación de métodos - Por inspección visual |

¿En que se basa un consensus docking?
|
° En candidatos con mejor puntaje
° En utilizar más de un método computacional ° En seleccionar mejores candidatos de múltiples métodos ° En incluir métodos gráficos y de visualización |
Sitio alostérico
|
Sitio en donde se unen sustancias que modifican la actividad enzimática las cuales son conocidas como efectores alostéricos que al cambiar la estructura de la enzima implica la posibilidad de activación o in activación de esa enzima.
|

¿Qué moléculas se deben tener prioridad y evaluar primero?
|
Las moléculas que se deben sintetizar y evaluar primero son las moléculas que presenten una mejor energía de interacción (afinidad calculada)
|
¿Cuál es el modo de unión en el complejo alfa-azarona- HMGR
|
Los tres oxígenos que se encuentran en la molécula forman puentes de hidrógeno cuando interaccionan con HMGR.
|

¿Cuáles son los objetivos principales del cribado virtual?
|
1. Incrementar la probabilidad de encontrar moléculas activas
2. Descubrir Hits computacionales 3. Descubrir Hits experimentales 4. Realizar diferentes evaluaciones exprimentales 5. Optimización (SAR local) y desarrollo |

¿Cuáles son los aspectos generales del "Screening virtual"?
|
Ya sea basado en el ligando o basado en la estructura, es un estudio computacional (screening "in silico") , en donde de varios compuestos seleccionados, se realiza un screening experimental, se obtienen los compuestos activos, se lleva a cabo su optimización y se realizan estudios se diseño "in silico"
|
¿De qué depende que se realice un acoplamiento molecular?
|
Depende de que se conozca la estructura en 3D del receptor (en caso de basado en la estructura).
|
¿De qué depende que se realice una similitud molecular?
|
Depende de que se conozcan los diferentes compuestos activos (en caso de basado en el ligando).
|
¿De qué depende que se realice un modelo del farmacóforo?
|
Este depende de que se conozca tanto la estructura en 3D del receptor como que se sepa cuales son los compuestos activos.
|

¿Qué es la epigenética?
|
Es el estudios de las modificaciones hereditarias en la cromatina que, sin modificar la secuencia de nucléotidos del DNA, regulan la expresión de genes.
|
En que consiste en cribado virtual de fármacos aprobados
|
Basados en la teoría de que moléculas similares tienes propiedades similares, se realiza un cribado virtual por similitud molecular entre una molécula en estudio y una biblioteca de fármacos aprobados ya reportada.
|
¿Cuáles son algunos conceptos en general del "Diseño de novo"?
|
° Que tiene como objetivo la automatización del diseño basado en la estructura
° Sugiere ideas de diferentes compuestos para su posterior síntesis ° Síntesis de moléculas pequeñas |

¿Cuáles son las etapas principales del diseño de novo?
|
1. Análisis del sitio de unión
2. Construcción de moléculas 3. Ordenamiento y selección de los mejores candidatos |

¿Cuál es la ventaja en el diseño en base a fragmentos?
|
Se espera que debido a que cada fragmento tiene un sitio de unión muy bueno, al final la molécula completa tenga aun mejor afinidad.
|
¿Cuales son algunas de las desventajas del diseño en base a fragmentos?
|
1. Que es necesaria la caracterización estructural de los mejores fragmentos y esto incluye más pasos experimentales además de costos
2. La unión de los fragmentos en algunas ocasiones puede llegar a ser algo complicada |
¿ En que puede consistir la biblioteca de los compuestos a usar para el diseño en base a fragmentos?
|
La biblioteca puede consistir en moléculas que provengan ya sea de productos naturales, de síntesis o en algunos casos computacionales
|
¿Cuál es la similitud entre el diseño de novo y el diseño en base a fragmentos?
|
Como similitud, ambos diseños tienen como finalidad que los dos buscan un mejor ajuste molecular.
|

¿Cuál es la principal diferencia entre el diseño de novo y el diseño en base a fragmentos?
|
Que el diseño de novo es un método computacional mientras que el diseño en base a fragmentos es un método experimental.
|